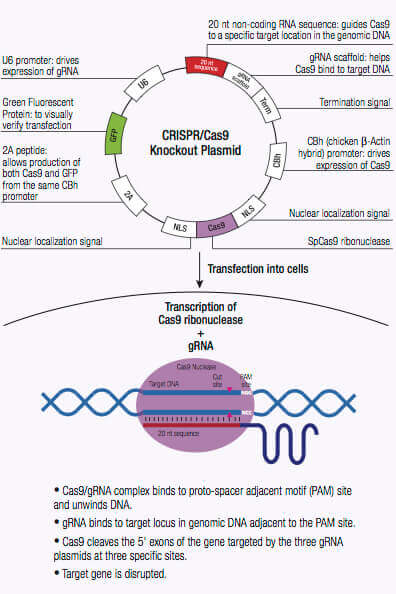
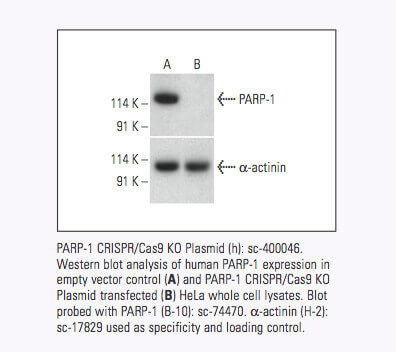
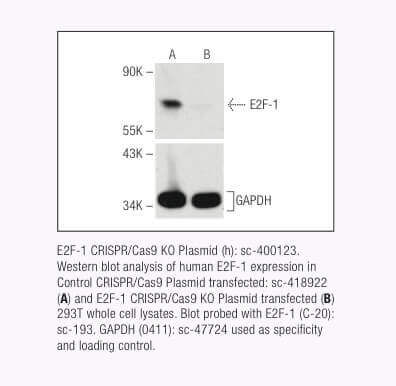
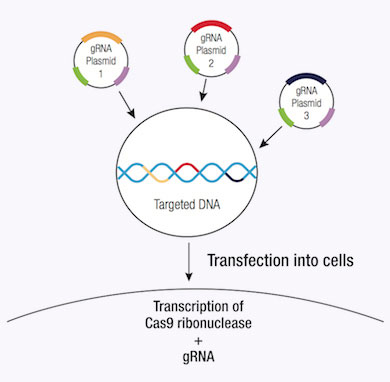
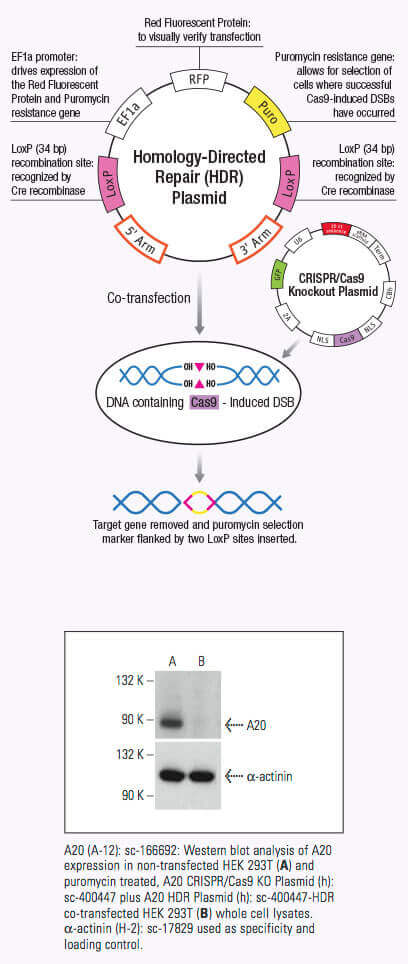
CRISPR Systems
산타크루즈 바이오테크놀로지는 현재 18,910개 이상의 인간 및 18,340개 이상의 마우스 단백질 암호화 유전자에 대한 표적 특이적 CRISPR/Cas9 녹아웃(KO) 플라스미드, CRISPR 이중 니카제 플라스미드, CRISPR/ dCas9 활성화 플라스미드 및 CRISPR 렌티 활성화 시스템을 제공하고 있습니다.
Quick Links
CRISPR/Cas9 역사
CRISPR/Cas시스템은 Archea와 박테리아의 고유 면역보호 시스템이 외부 유전물질을 분쇄하는 원리를 이용한시스템이다. 이런 유기체들은 살균바이러스의 외부유전물질을 획득하여 CRISPR loci (1,2)에 통합시킨다. 이 소재는 spacer로도 얄려져 있으며 sequence-specific fragment를 만들어 미래의 살균바이러스의 감염에 대항한다. 이런 sequence-specific fragments는 short CRISPR RNAs (crRNAs)로 번역되여 외부침입상보DNA의 직접적인 cleavage를 가이드하는데 이역시 CRISPR loci (1,2)로 encod된 CRISPR-associated (Cas) 프로틴의 뉴클리아제활성을 통해 완성되고 있다. Cas9뉴클리아제는 type II CRISPR시스템으로서 RNA바인딩구역, alpha helix recognition lobe (REC), DNA cleavage를 위한 RuvC 와 HNH 를 포함한 nuclease lobe 와 protospacer adjacent motif (PAM) 상호작용 사이트 (1,2)를 포함하고있다. crRNA는 REC lobe내부의 bridge helix와 결합하여 Cas9뉴클리아제와 복합물을 형성하며 crRNA (1,2,3)의 backbone와 멀티플 염다리를 형성합니다.
crRNA가 Cas9와 결합하면 Cas9 nuclease의 conformation이 변화하여 DNA바인딩 채널(1,2,3)을 만듭니다. Cas9/crRNA복합물은 DNA의 PAM (5'-NGG) 사이트를 (4,5,6) 스캔해냅니다. PAM s사이트의 인지는 DNA의 unwinding하여 crRNA 가 PAM사이트 주변의 complementary DNA을 체크하도록 리드합니다. Cas9가 PAM site주변의crRNA와 상보적인 DNA sequence와 바인딩하면, REC lobe내부의 bridge helix가 target DNA (3,4,7)와 RNA-DNA heteroduplex를 이루게 됩니다. PAM사이트 인지는 타깃 DNA의 double-stranded break (DSB)를 초래하는 nucleolytic HNH and RuvC domains의 activating에 참여해 DNA degradation (1,2,5,8)을 리드합니다. crRNA와 상보적이지 않을경우 Cas9 는 풀려나와 다른 PAM사이트를 찾게 됩니다(7). 타깃 유전자의 DNA 사슬이 끊기면 nonhomologous end-joining (NHEJ) repair pathway를 통해 복구되는데 이는 삽입이나 삭제를 초래하여 오류가 발생합니다, 반면 homologous directed repair (HDR) pathway를 통해 복구할경우, 유전자의 특정위치에 selected markers를 재결합시킵니다 (2,9,10). CRISPR/Cas9 mechanism은 포유류 세포를 포함한 여러시스템의 genomic engineering에 널리 사용될수있습니다.
DSBs를 형성하는 유전자 교정은 징크핑거뉴클리아제 (ZFN), 탈렌(TALENs) 등 DNA 인지 가능한 메가뉴클리아제로 진행하지만 모두 제한성을 보이고 있습니다. 메가뉴클리아제를 사용할시 뉴클리아제와 DNA사이의 특정된 위치의 인지가 어렵습니다(2). 이외에 징크핑거뉴클리아제와 탈렌은 디자인이 어려우며 3nt의 DNA를 인지하기 어렵습니다 (2). 싱글가이드 RNA (sgRNAs)는 crRNAs와 같이 디자인이 쉬우며 Cas9 뉴클리아제와 같은 벡터로 발현하여 타깃 유전자 교정을 진행할수 있습니다. CRISPR/Cas 9 시스템은 민감성이 뛰여나며 심사시 small hairpin RNAs보다 유효성이 뛰여납니다.
CRISPR/Cas9 시스템의 중요한 이점은 게놈 DNA에 유도하는 DSB의 높은 효율에 있습니다. 이 효율은 off-target의 영향을 받아 CRISPR/Cas9의 편집특이성을 감소시킬수 있습니다. 이 특이성은 CRISPR double nickase system으로 향상가능한데 이는 한쌍의 plasmid로 제공하고 있으며 매 plasmid마다 Cas9 (D10A) nickase mutant (Cas9n)를 인코딩하며 target-specific guide RNA (12)에 의하며 타깃의 특정구역에 유도됩니다. 매개의 Cas9n/sgRNA complex는 guide RNA (12)의 상보DNA strand에 단하나의 nick를 발생합니다. 매개의 guide RNA는 약 20bp가량 떨어진거리에서 opposite strands의 타깃sequence를 식별합니다. 한쌍의Cas9n/sgRNA complexes가 편집한 더블nick는 DSB를 모방합니다(12). 이처럼 한쌍의 guide RNA를 이용하여 Cas9(매개)유전자 편집의 특이성을 증가함과 동시에 높은 효율을 유지합니다 (12).
뿐만아니라 지놈편집에는 CRISPR system으로 내인성유전자(endogenous gene)의 활성화 강화를 실현할수 있습니다(13). CRISPR system을 부분적으로 개조하여 synergistic activation mediator (SAM) complex를 형성하면 고효율적인 specific transcription activation system (13)을 얻을수 있습니다. Cas9뉴클리아제를 개조하여 SAM complex의 한부분을 만듭니다. Cas9의 the catalytic domains을 deactivated시켜 dCas9을 얻어 transcription activation domain (VP64)과 융합시킵니다. target specific guide RNA (sgRNA)로 유도된 dCas9-VP64-sgRNA complex는 내인성유전자의 Transcriptional Start Site (TSS) 의 -200 bp region에 타깃을 맞추어 유전자발현을 upregulate합니다(13). To further enhance transcription, the sgRNA has been modified by appending a minimal hairpin aptamer to the tetraloop and stem loop 2 (13). 이 앱타머는 선택적으로dimerized MS2 bacteriophage coat proteins을 바인딩합니다 (13). MS2 프로틴에 p65 와 HSF1 transactivation domains을 융합하면 MS2-P65-HSF1 fusion protein을 형성하는데 이는 transcription factors를 불러드리는 반응을 강화하여dCas9-mediated gene activation 효율을 증가합니다(13). Activation제품은 모든 cell타입의SAM Transcription Activation System 의 delivery효율보장을 위하여 위한형질주입가능한 standard plasmid와 형질도입가능한 plasmid가 packing된 lentiviral제품을 제공합니다.
Quick Links
CRISPR/Cas9-directed Double Strand Break (DSB)