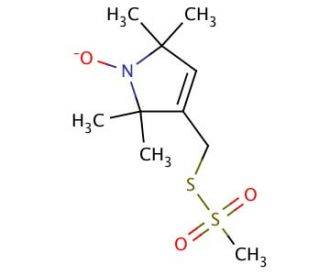
Structure moléculaire de MTSL, Numéro CAS: 81213-52-7
MTSL (CAS 81213-52-7)
Voir les citations produits (56)
Noms alternatifs:
MTSL also known as (1-Oxyl-2,2,5,5-tetramethylpyrroline-3- methyl)methanethiosulfonate; MTS; MTSSL; Otmpmms; Otpm-mts; 2,5-Dihydro-2,2,5,5-tetramethyl-3-[[(methylsulfonyl)thio]methyl]-1H-pyrrol-1-yloxy; (1-Oxyl-2,2,5,5-tetramethyl-delta(3)-pyrroline-3-methyl)methanethiosulfonate; (2,2,5,5-tetramethyl-3-{[(methylsulfonyl)sulfanyl]methyl}-2,5-dihydro-1H-pyrrol-1-yl)oxidanyl
Application(s):
MTSL est un spin-label hautement réactif, spécifique aux thiols, qui constitue une sonde conformationnelle spécifique de la structure du site thiol.
Numéro CAS:
81213-52-7
Pureté:
≥98%
Masse Moléculaire:
264.38
Formule Moléculaire:
C10H18NO3S2
Pour la Recherche Uniquement. Non conforme pour le Diagnostic ou pour une Utilisation Thérapeutique.
* Consulter le Certificat d'Analyses pour les données spécifiques à un lot (incluant la teneur en eau).
ACCÈS RAPIDE AUX LIENS
Informations pour la commande
Références bibliographiques
Description
Information Technique
Données de Sécurité
SDS & Certificat d’Analyses
Le MTSL est un marqueur de spin hautement réactif et spécifique aux thiols. Le MTSL est une sonde conformationnelle spécifique de la structure du site thiol grâce à sa liberté de rotation minimale et à sa distance par rapport à la liaison disulfure covalente avec la macromolécule étudiée. La MTSL peut être utilisée pour marquer les résidus cystéine dans les protéines, le marquage dirigé par site et le marquage SDS. MTSL permet de déterminer la structure et la dynamique des protéines et d'étudier les interactions protéine-protéine et protéine-oligonucléotide.
MTSL (CAS 81213-52-7) Références
- Artéfacts potentiels dans l'utilisation d'un système de protéine de fusion de la glutathion S-transférase et de méthodes de résonance paramagnétique électronique de marquage de spin pour étudier les interactions protéine-protéine. | Antoniou, C. and Fung, LW. 2008. Anal Biochem. 376: 160-2. PMID: 18316034
- Conformation de l'état de canal fermé de la colicine A dans les protéoliposomes: un modèle parapluie. | Padmavathi, PV. and Steinhoff, HJ. 2008. J Mol Biol. 378: 204-14. PMID: 18353363
- Conformation et position membranaire de la région reliant les deux domaines C2 de la synaptotagmine 1 par marquage de spin dirigé. | Huang, H. and Cafiso, DS. 2008. Biochemistry. 47: 12380-8. PMID: 18956883
- Nouveaux aperçus du rôle du domaine N-terminal désordonné de WIP révélés par la caractérisation structurale par RMN. | Elazari-Shalom, H., et al. 2015. FEBS J. 282: 700-14. PMID: 25495558
- Une approche combinée de RMN et de calcul pour étudier la liaison de peptides à une protéine répétée d'Armadillo. | Ewald, C., et al. 2015. J Mol Biol. 427: 1916-33. PMID: 25816772
- Étude structurale et biophysique de l'interaction d'un domaine SH2 mutant de Grb2 (W121G) avec son phosphopeptide connu. | Papaioannou, D., et al. 2016. Protein Sci. 25: 627-37. PMID: 26645482
- Mesures de distance dans la protéine peridinine-chlorophylle-a par spectroscopie PELDOR induite par la lumière. Analyse de la localisation de l'état triplet. | Di Valentin, M., et al. 2016. Biochim Biophys Acta. 1857: 1909-1916. PMID: 27659505
- Régularisation optimale de Tikhonov pour la spectroscopie DEER. | Edwards, TH. and Stoll, S. 2018. J Magn Reson. 288: 58-68. PMID: 29414064
- Marquage des nucléotides pour la spectroscopie ESR des protéines liant l'ATP. | Muok, AR., et al. 2018. Appl Magn Reson. 49: 1385-1395. PMID: 30686862
- Un commutateur moléculaire régulant la répression et l'activation transcriptionnelles de PPARγ. | Shang, J., et al. 2020. Nat Commun. 11: 956. PMID: 32075969
- La préformation structurelle à longue portée de la protéine associée au oui précède la formation d'un complexe de rencontre avec TEAD. | Feichtinger, M., et al. 2022. iScience. 25: 104099. PMID: 35378854
Informations pour la commande
| Nom du produit | Ref. Catalogue | COND. | Prix HT | QTÉ | Favoris | |
MTSL, 10 mg | sc-208677 | 10 mg | $170.00 | |||
MTSL, 50 mg | sc-208677A | 50 mg | $400.00 |
