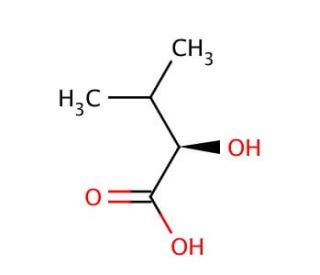
D-α-Hydroxyisovaleric acid (CAS 17407-56-6)
ACCÈS RAPIDE AUX LIENS
L'acide D-α-Hydroxyisovalérique, appelé acide (R)-2-hydroxyisobutyrique (RHIBA), est un composé organique que l'on trouve à l'état naturel dans une grande variété de plantes, d'animaux et de micro-organismes. L'acide D-α-Hydroxyisovalérique possède une chiralité, provenant d'un atome de carbone asymétrique solitaire, et joue un rôle important en tant qu'intermédiaire dans la biosynthèse des acides aminés, des acides gras et de divers autres composés. Les applications de l'acide D-α-Hydroxyisovalérique dans la recherche scientifique sont nombreuses. Il sert de substrat pour la synthèse de divers composés, notamment des acides aminés, des acides gras et d'autres métabolites. En outre, il est utilisé comme réactif analytique, facilitant la détermination des niveaux de concentration de divers composés. En outre, l'acide D-α-Hydroxyisovalérique joue un rôle important dans les études de biochimie et de biologie moléculaire, notamment dans l'étude de la structure et de la fonction des enzymes. Il participe en tant que substrat au cycle de Krebs, une série de réactions chimiques qui génèrent de l'énergie cellulaire. En outre, dans la signalisation cellulaire, l'acide D-α-Hydroxyisovalérique joue un rôle dans la génération d'adénosine monophosphate cyclique (AMPc), une molécule de signalisation vitale qui régule de nombreux processus cellulaires.
D-α-Hydroxyisovaleric acid (CAS 17407-56-6) Références
- Déplacements chimiques 13C RMN des formes cristallines triclinique et monoclinique de la valinomycine. | Kameda, T., et al. 2004. J Biomol NMR. 29: 281-8. PMID: 15213426
- Décryptage des codes de biosynthèse du puissant cyclodepsipeptide valinomycine anti-SARS-CoV chez Streptomyces tsusimaensis ATCC 15141. | Cheng, YQ. 2006. Chembiochem. 7: 471-7. PMID: 16511823
- Études structurales sur les enniatines mineures de Fusarium sp. VI 03441: nouvelles enniatines contenant de la N-méthyl-thréonine. | Uhlig, S., et al. 2009. Toxicon. 53: 734-42. PMID: 19249325
- Biosynthèse reconstituée de l'antibiotique macrolactone nonribosomique valinomycine chez Escherichia coli. | Jaitzig, J., et al. 2014. ACS Synth Biol. 3: 432-8. PMID: 24350980
- Un nouvel antibiotique à base d'enniatine provenant de l'endophyte Fusarium tricinctum Corda. | Zaher, AM., et al. 2015. J Antibiot (Tokyo). 68: 197-200. PMID: 25315756
- Production accrue de valinomycine chez des mutants de Streptomyces sp. M10 défectueux dans la biosynthèse de la bafilomycine et l'expression du complexe α-cétoacide déshydrogénase à chaîne ramifiée. | Lee, DW., et al. 2015. J Ind Microbiol Biotechnol. 42: 1507-17. PMID: 26335568
- Cyclodepsipeptides antiprolifératifs de l'actinomycète marin Streptomyces sp. P11-23B réduisant les enzymes métaboliques tumorales de la glycolyse, de la glutaminolyse et de la lipogenèse. | Ye, X., et al. 2017. Phytochemistry. 135: 151-159. PMID: 28049552
- Trapping biosynthetic acyl-enzyme intermediates with encoded 2,3-diaminopropionic acid (piégeage des intermédiaires acyl-enzymes biosynthétiques avec de l'acide 2,3-diaminopropionique codé). | Huguenin-Dezot, N., et al. 2019. Nature. 565: 112-117. PMID: 30542153
- Synthèse de la beauvéricine par une enzyme multifonctionnelle. | Peeters, H., et al. 1988. J Antibiot (Tokyo). 41: 352-9. PMID: 3366693
- Le peptide non ribosomal valinomycine: De la découverte à la bioactivité et à la biosynthèse. | Huang, S., et al. 2021. Microorganisms. 9: PMID: 33917912
- La modélisation métabolique à l'échelle du génome combinée au profilage du transcriptome permet de comprendre le mécanisme du métabolisme de Streptococcus thermophilus CH8. | Rau, MH., et al. 2022. Appl Environ Microbiol. 88: e0078022. PMID: 35924931
- Stéréocomplexation entre oligomères substitués de type PLA et influence sur la dégradation hydrolytique | Andersson, S. R., Hakkarainen, M., & Albertsson, A. C. 2013. Polymer. 54(16): 4105-4111.
Informations pour la commande
| Nom du produit | Ref. Catalogue | COND. | Prix HT | QTÉ | Favoris | |
D-α-Hydroxyisovaleric acid, 1 g | sc-239638 | 1 g | $158.00 |
