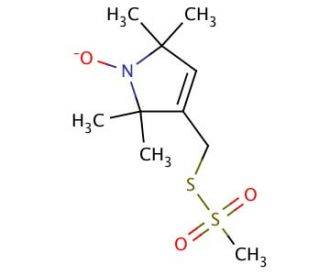
Molekülstruktur von MTSL, CAS-Nummer: 81213-52-7
MTSL (CAS 81213-52-7)
Produktreferenzen ansehen (56)
Alternative Namen:
MTSL also known as (1-Oxyl-2,2,5,5-tetramethylpyrroline-3- methyl)methanethiosulfonate; MTS; MTSSL; Otmpmms; Otpm-mts; 2,5-Dihydro-2,2,5,5-tetramethyl-3-[[(methylsulfonyl)thio]methyl]-1H-pyrrol-1-yloxy; (1-Oxyl-2,2,5,5-tetramethyl-delta(3)-pyrroline-3-methyl)methanethiosulfonate; (2,2,5,5-tetramethyl-3-{[(methylsulfonyl)sulfanyl]methyl}-2,5-dihydro-1H-pyrrol-1-yl)oxidanyl
Anwendungen:
MTSL ist ein hochreaktives, thiolspezifisches Spinlabel, das eine spezifische Konformationssonde der Thiolstellenstruktur ist
CAS Nummer:
81213-52-7
Reinheit:
≥98%
Molekulargewicht:
264.38
Summenformel:
C10H18NO3S2
Ausschließlich für Forschungszwecke. Nicht Geeignet für Verwendung in Diagnostik oder Therapie.
* Schauen Sie auf das Analysezertifikat (CoA), um die genauen Daten (inkl. Wassergehalt) Ihrer Produktionscharge (Lot) zu sehen.
Direktverknüpfungen
Bestellinformation
Publikationen
Beschreibung
Hintergrundinformationen
Sicherheitshinweis
SDS (SDB) & Analysenzertifikate
MTSL ist ein hochreaktiver, thiol-spezifisches Spin-Label. MTSL ist eine spezifische konformationelle Sonde für die Struktur von Thiolstellen, aufgrund seiner minimalen Rotationsfreiheit und Entfernung von der kovalenten Disulfidverbindung zu der zu untersuchenden Makromolekül. MTSL kann verwendet werden, um Cysteinresten in Proteinen zu markieren, gezielte Markierung und SDS-Markierung durchzuführen. MTSL ermöglicht die Bestimmung der Proteinstruktur und Proteindynamik sowie die Untersuchung von Protein-Protein- und Protein-Oligonukleotid-Wechselwirkungen.
MTSL (CAS 81213-52-7) Literaturhinweise
- Potenzielle Artefakte bei der Verwendung eines Glutathion-S-Transferase-Fusionsproteinsystems und von Spin-Labeling-Methoden der elektronenparamagnetischen Resonanz zur Untersuchung von Protein-Protein-Wechselwirkungen. | Antoniou, C. and Fung, LW. 2008. Anal Biochem. 376: 160-2. PMID: 18316034
- Konformation des geschlossenen Kanalzustands von Colicin A in Proteoliposomen: ein Regenschirmmodell. | Padmavathi, PV. and Steinhoff, HJ. 2008. J Mol Biol. 378: 204-14. PMID: 18353363
- Konformation und Membranposition der Region, die die beiden C2-Domänen in Synaptotagmin 1 verbindet, durch ortsgerichtete Spinmarkierung. | Huang, H. and Cafiso, DS. 2008. Biochemistry. 47: 12380-8. PMID: 18956883
- Neue Erkenntnisse über die Rolle der ungeordneten N-terminalen WIP-Domäne durch NMR-Strukturcharakterisierung. | Elazari-Shalom, H., et al. 2015. FEBS J. 282: 700-14. PMID: 25495558
- Ein kombinierter NMR- und Berechnungsansatz zur Untersuchung der Peptidbindung an ein entworfenes Armadillo-Repeat-Protein. | Ewald, C., et al. 2015. J Mol Biol. 427: 1916-33. PMID: 25816772
- Strukturelle und biophysikalische Untersuchung der Interaktion einer mutierten Grb2 SH2-Domäne (W121G) mit ihrem kognitiven Phosphopeptid. | Papaioannou, D., et al. 2016. Protein Sci. 25: 627-37. PMID: 26645482
- Abstandsmessungen in Peridinin-Chlorophyll-A-Protein durch lichtinduzierte PELDOR-Spektroskopie. Analyse der Triplett-Zustandslokalisierung. | Di Valentin, M., et al. 2016. Biochim Biophys Acta. 1857: 1909-1916. PMID: 27659505
- Optimale Tikhonov-Regularisierung für DEER-Spektroskopie. | Edwards, TH. and Stoll, S. 2018. J Magn Reson. 288: 58-68. PMID: 29414064
- Nukleotid-Spinmarkierung für die ESR-Spektroskopie von ATP-bindenden Proteinen. | Muok, AR., et al. 2018. Appl Magn Reson. 49: 1385-1395. PMID: 30686862
- Ein molekularer Schalter, der die transkriptionelle Unterdrückung und Aktivierung von PPARγ reguliert. | Shang, J., et al. 2020. Nat Commun. 11: 956. PMID: 32075969
- Eine weitreichende strukturelle Vorbildung im yes-assoziierten Protein geht der Bildung eines Begegnungskomplexes mit TEAD voraus. | Feichtinger, M., et al. 2022. iScience. 25: 104099. PMID: 35378854
Bestellinformation
| Produkt | Katalog # | EINHEIT | Preis | ANZAHL | Favoriten | |
MTSL, 10 mg | sc-208677 | 10 mg | $170.00 | |||
MTSL, 50 mg | sc-208677A | 50 mg | $400.00 |
