Subscrever / Inscrever-se Conta
Corrinho de compra (0)
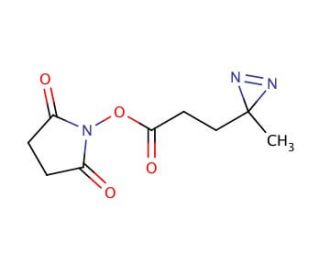
Estrutura molecular do SDA (NHS-Diazirine)
SDA (NHS-Diazirine)
Nomes alternativos:
Succinimidyl 4,4′-azipentanoate
Aplicacao:
SDA (NHS-Diazirine) é um reticulador fotorreactivo à base de NHS-diazirina. Permeável à membrana e clivável com um braço espaçador de 3,9 Angstrom.
Peso Molecular:
225.20
Separar por Funcao:
C9H11N3O4
Para uso em exclusivo em pesquisa. Não se destina a uso em diagnostico e tratamento.
* Refere-se a Certificado de Análise para data especifica de lotes (incluindo-se o conteúdo de agua).
LINKS RÁPIDOS
Informacoes sobre ordens
Descricao
Informações Técnicas
Informações de Segurança
SDS & Certificado de Análise
O SDA (NHS-Diazirina) é um reticulador heterobifuncional amina e fotorreactivo, permeável à membrana, com um braço espaçador de 3,9 Angstrom. O grupo fotorreactivo diazirina tem uma melhor fotoestabilidade do que as aril azidas, mas é mais eficazmente ativado por luz UV de onda longa (330-370nm). A ativação da diazirina liberta azoto para produzir um intermediário carbeno altamente reativo, que se liga indiscriminadamente a um grupo funcional próximo.
SDA (NHS-Diazirine) Referencias
- Claudin-2 forma homodímeros e é um componente de um complexo proteico de elevado peso molecular. | Van Itallie, CM., et al. 2011. J Biol Chem. 286: 3442-50. PMID: 21098027
- O KCNE1 induz a fenestração no complexo do canal Kv7.1/KCNE1, o que permite uma seleção farmacológica altamente específica. | Wrobel, E., et al. 2016. Nat Commun. 7: 12795. PMID: 27731317
- Sondagem da interface de interação dos complexos GADD45β/MKK7 e MKK7/DTP3 por espetrometria de massa de reticulação química. | Rega, C., et al. 2018. Int J Biol Macromol. 114: 114-123. PMID: 29572137
- PLiMAP: Marcação baseada na proximidade de proteínas associadas à membrana. | Jose, GP. and Pucadyil, TJ. 2020. Curr Protoc Protein Sci. 101: e110. PMID: 32603530
- Controlo da absorção, libertação e erosão de fármacos em hidrogéis com engenharia de proteínas fotopadronizadas. | Wang, Y., et al. 2020. Biomacromolecules. 21: 3608-3619. PMID: 32786534
- A análise das redes de ARN-proteínas com RNP-MaP define centros funcionais no ARN. | Weidmann, CA., et al. 2021. Nat Biotechnol. 39: 347-356. PMID: 33077962
- Hidrogéis de polímeros proteicos de ligação cruzada de pé livre para libertação sustentada de fármacos. | Wang, Y., et al. 2021. Biomacromolecules. 22: 1509-1522. PMID: 33685120
- Variantes não codificantes no cancro: Mechanistic Insights and Clinical Potential for Personalized Medicine (Percepções Mecânicas e Potencial Clínico para a Medicina Personalizada). | Lange, M., et al. 2021. Noncoding RNA. 7: PMID: 34449663
- Efeito de Catiões Metálicos Divalentes na Conformação, Comportamento Elástico e Libertação Controlada de um Hidrogel de Engenharia de Proteínas Fotocrosslinked. | Wang, Y., et al. 2021. ACS Appl Bio Mater. 4: 3587-3597. PMID: 35014444
- O envolvimento cooperativo e a subsequente deslocação selectiva das proteínas SR definem o andaime estrutural 3D do pré-RNAm para a montagem inicial do spliceossoma. | Saha, K. and Ghosh, G. 2022. Nucleic Acids Res. 50: 8262-8278. PMID: 35871302
- O SpotLink permite a identificação sensível e precisa de ligações cruzadas não específicas do local à escala do proteoma. | Zhang, W., et al. 2022. Brief Bioinform. 23: PMID: 36093786
- Sondagem química correlacionada de molécula única: Uma revolução na análise da estrutura do ARN. | Mustoe, AM., et al. 2023. Acc Chem Res. 56: 763-775. PMID: 36917683
- Identificação estrutural de elementos de ARN que regulam a arquitetura e a replicação do genoma do vírus da dengue. | Boerneke, MA., et al. 2023. Proc Natl Acad Sci U S A. 120: e2217053120. PMID: 37011200
Informacoes sobre ordens
| Nome do Produto | Numero de Catalogo | UNID | Preco | Qde | FAVORITOS | |
SDA (NHS-Diazirine), 50 mg | sc-397278 | 50 mg | $437.00 |
