Subscrever / Inscrever-se Conta
Corrinho de compra (0)
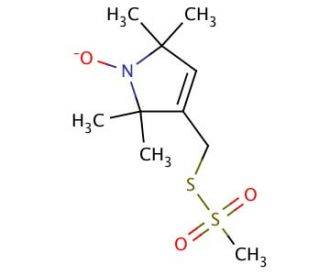
Estrutura molecular do MTSL, Número CAS: 81213-52-7
MTSL (CAS 81213-52-7)
Veja citações de produtos (56)
Nomes alternativos:
MTSL also known as (1-Oxyl-2,2,5,5-tetramethylpyrroline-3- methyl)methanethiosulfonate; MTS; MTSSL; Otmpmms; Otpm-mts; 2,5-Dihydro-2,2,5,5-tetramethyl-3-[[(methylsulfonyl)thio]methyl]-1H-pyrrol-1-yloxy; (1-Oxyl-2,2,5,5-tetramethyl-delta(3)-pyrroline-3-methyl)methanethiosulfonate; (2,2,5,5-tetramethyl-3-{[(methylsulfonyl)sulfanyl]methyl}-2,5-dihydro-1H-pyrrol-1-yl)oxidanyl
Aplicacao:
MTSL é um marcador de spin altamente reativo e específico do tiol, que constitui uma sonda conformacional específica da estrutura do local do tiol
Numero VAT:
81213-52-7
Privada:
≥98%
Peso Molecular:
264.38
Separar por Funcao:
C10H18NO3S2
Para uso em exclusivo em pesquisa. Não se destina a uso em diagnostico e tratamento.
* Refere-se a Certificado de Análise para data especifica de lotes (incluindo-se o conteúdo de agua).
LINKS RÁPIDOS
Informacoes sobre ordens
Citacoes dos Produtos
Descricao
Informações Técnicas
Informações de Segurança
SDS & Certificado de Análise
O MTSL é um spin-label altamente reativo e específico para tióis. O MTSL é uma sonda conformacional específica da estrutura do local do tiol devido à sua liberdade rotacional mínima e à distância da ligação covalente de dissulfeto à macromolécula em estudo. A MTSL pode ser usada para marcar resíduos de cisteína em proteínas, marcação direcionada ao local e marcação SDS. O MTSL permite a determinação da estrutura e da dinâmica das proteínas e o estudo das interações proteína-proteína e proteína-oligonucleotídeo.
MTSL (CAS 81213-52-7) Referencias
- Potenciais artefactos na utilização de um sistema de proteína de fusão de glutationa S-transferase e métodos de ressonância paramagnética eletrónica de marcação de spin para estudar interações proteína-proteína. | Antoniou, C. and Fung, LW. 2008. Anal Biochem. 376: 160-2. PMID: 18316034
- Conformação do estado de canal fechado da colicina A em proteolipossomas: um modelo de umbrella. | Padmavathi, PV. and Steinhoff, HJ. 2008. J Mol Biol. 378: 204-14. PMID: 18353363
- Conformação e posição membranar da região que liga os dois domínios C2 na sinaptotagmina 1 por marcação de spin dirigida ao local. | Huang, H. and Cafiso, DS. 2008. Biochemistry. 47: 12380-8. PMID: 18956883
- Novos conhecimentos sobre o papel do domínio N-terminal desordenado do WIP revelados pela caraterização estrutural por RMN. | Elazari-Shalom, H., et al. 2015. FEBS J. 282: 700-14. PMID: 25495558
- Uma abordagem combinada de RMN e computacional para investigar a ligação de péptidos a uma proteína de repetição Armadillo concebida. | Ewald, C., et al. 2015. J Mol Biol. 427: 1916-33. PMID: 25816772
- Investigação estrutural e biofísica da interação de um domínio SH2 mutante da Grb2 (W121G) com o seu fosfopeptídeo cognato. | Papaioannou, D., et al. 2016. Protein Sci. 25: 627-37. PMID: 26645482
- Medidas de distância na proteína a peridinina-clorofila por espetroscopia PELDOR induzida por luz. Análise da localização do estado tripleto. | Di Valentin, M., et al. 2016. Biochim Biophys Acta. 1857: 1909-1916. PMID: 27659505
- Regularização óptima de Tikhonov para espetroscopia DEER. | Edwards, TH. and Stoll, S. 2018. J Magn Reson. 288: 58-68. PMID: 29414064
- Marcação de nucleótidos para espetroscopia ESR de proteínas de ligação a ATP. | Muok, AR., et al. 2018. Appl Magn Reson. 49: 1385-1395. PMID: 30686862
- Um interrutor molecular que regula a repressão e ativação transcricional do PPARγ. | Shang, J., et al. 2020. Nat Commun. 11: 956. PMID: 32075969
- A pré-formação estrutural de longo alcance na proteína associada a yes precede a formação do complexo de encontro com TEAD. | Feichtinger, M., et al. 2022. iScience. 25: 104099. PMID: 35378854
Informacoes sobre ordens
| Nome do Produto | Numero de Catalogo | UNID | Preco | Qde | FAVORITOS | |
MTSL, 10 mg | sc-208677 | 10 mg | $170.00 | |||
MTSL, 50 mg | sc-208677A | 50 mg | $400.00 |
