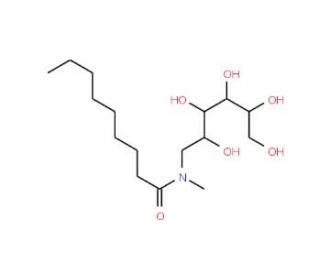
MEGA-9 (CAS 85261-19-4)
LINKS RÁPIDOS
MEGA-9 é um surfactante utilizado em aplicações de investigação que requerem a solubilização de compostos hidrofóbicos. As suas propriedades anfifílicas tornam-no adequado para a criação de micelas em soluções aquosas, o que é particularmente útil no estudo de proteínas de membrana e das suas interacções com lípidos. O MEGA-9 é também utilizado na preparação de bicamadas lipídicas e lipossomas para ensaios biofísicos e bioquímicos. Em química analítica, o MEGA-9 é utilizado como catalisador de transferência de fase para a extração de moléculas orgânicas de fases aquosas para fases orgânicas, facilitando a purificação e a análise destes compostos. Além disso, este surfactante é utilizado na formulação de detergentes para a limpeza suave de materiais biológicos num ambiente laboratorial.
MEGA-9 (CAS 85261-19-4) Referencias
- Auto-agregação de MEGA-9 (N-nonanoil-N-metil-D-glucamina) em meio aquoso: físico-química dos comportamentos interfaciais e em solução, com especial referência à energia de formação e ao microambiente micelar. | Pan, A., et al. 2013. J Phys Chem B. 117: 7578-92. PMID: 23718221
- Cristalização e análise cristalográfica preliminar de raios X de uma putativa sintase de péptido não ribossómico AmbB de Pseudomonas aeruginosa. | Wang, Y., et al. 2014. Acta Crystallogr F Struct Biol Commun. 70: 339-42. PMID: 24598922
- Avaliação da semelhança de sequências de ADN com base em padrões frequentes e entropia. | Xie, X., et al. 2015. BMC Genomics. 16 Suppl 3: S5. PMID: 25707937
- Correção: Activity of the Human Rhinovirus 3C Protease Studied in Various Buffers, Additives and Detergents Solutions for Recombinant Protein Production [Atividade da Protease 3C do Rinovírus Humano Estudada em Vários Tampões, Aditivos e Detergentes para a Produção de Proteínas Recombinantes]. | Ullah, R., et al. 2016. PLoS One. 11: e0160128. PMID: 27442507
- Sequências completas do genoma de estirpes simpátricas recombinantes e não recombinantes do Rice yellow mottle virus do Quénia Ocidental. | Adego, AK., et al. 2018. Genome Announc. 6: PMID: 29472342
- Genómica comparativa de estirpes de vacinas contra a tosse convulsa de células inteiras da Índia. | Alai, S., et al. 2020. BMC Genomics. 21: 345. PMID: 32381023
- Calibração, correção e aplicação do sistema de imagiologia no domínio da frequência espacial para a deteção de danos na superfície de pêras. | Luo, Y., et al. 2021. Foods. 10: PMID: 34574261
- Análise exaustiva da metalo-β-lactamase do tipo imipenemase (IMP): Uma distribuição global que ameaça a Ásia. | Pongchaikul, P. and Mongkolsuk, P. 2022. Antibiotics (Basel). 11: PMID: 35203838
- Síntese biocatalítica num só passo de surfactantes sustentáveis através da formação selectiva de ligações amida. | Lubberink, M., et al. 2022. Angew Chem Int Ed Engl. 61: e202205054. PMID: 35595679
- Um detetor de fluorescência altamente integrado e diminuto para testes no local de atendimento: Conceção e implementação de circuitos de processamento fotoelétrico e de acionamento de díodos emissores de luz (LED) com realimentação negativa dupla. | Wang, Y., et al. 2022. Biosensors (Basel). 12: PMID: 36140149
- Um módulo de fluorescência de canal duplo miniaturizado e integrado para PCR multiplex em tempo real no sistema portátil de deteção de ácidos nucleicos. | Fang, Y., et al. 2022. Front Bioeng Biotechnol. 10: 996456. PMID: 36172017
- Identificação de toda a família de genes DUF668 no genoma e análise da expressão sob stress de seca e sal em batata-doce [Ipomoea batatas (L.) Lam]. | Liu, E., et al. 2023. Genes (Basel). 14: PMID: 36672958
- Análise evolutiva da família de genes GH3 do melão (Cucumis melo L.) e identificação de genes GH3 relacionados com o crescimento e desenvolvimento do fruto. | Chen, S., et al. 2023. Plants (Basel). 12: PMID: 36987071
Informacoes sobre ordens
| Nome do Produto | Numero de Catalogo | UNID | Preco | Qde | FAVORITOS | |
MEGA-9, 1 g | sc-280958 | 1 g | $87.00 |
