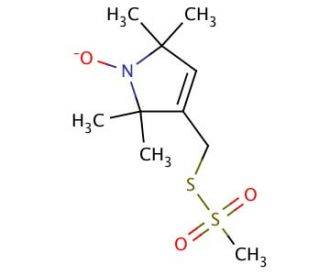
Struttura molecolare di MTSL, Numero CAS: 81213-52-7
MTSL (CAS 81213-52-7)
Vedere citazioni di prodotto (56)
Nomi alternativi:
MTSL also known as (1-Oxyl-2,2,5,5-tetramethylpyrroline-3- methyl)methanethiosulfonate; MTS; MTSSL; Otmpmms; Otpm-mts; 2,5-Dihydro-2,2,5,5-tetramethyl-3-[[(methylsulfonyl)thio]methyl]-1H-pyrrol-1-yloxy; (1-Oxyl-2,2,5,5-tetramethyl-delta(3)-pyrroline-3-methyl)methanethiosulfonate; (2,2,5,5-tetramethyl-3-{[(methylsulfonyl)sulfanyl]methyl}-2,5-dihydro-1H-pyrrol-1-yl)oxidanyl
Applicazione:
MTSL è una spin-label altamente reattiva e specifica per i tioli che costituisce una sonda conformazionale specifica della struttura del sito tiolico
Numero CAS:
81213-52-7
Purezza:
≥98%
Peso molecolare:
264.38
Formula molecolare:
C10H18NO3S2
Solo per uso in Ricerca. Non previsto per Uso Diagnostico o Terapeutico.
* Vedere Certificato di Analisi per informazioni sul lotto specifico (incluso il contenuto d'acqua).
LINK RAPIDI
Informazioni ordini
Citazioni prodotti
Descrizione
Informazione tecnica
Informazioni di sicurezza
SDS & Certificato d'analisi
L'MTSL è una spin-label altamente reattiva e specifica per i tioli. L'MTSL è una sonda conformazionale specifica della struttura del sito tiolico grazie alla sua minima libertà rotazionale e alla distanza dal legame disolfuro covalente con la macromolecola da studiare. L'MTSL può essere utilizzato per etichettare i residui di cisteina nelle proteine, per l'etichettatura diretta al sito e per l'etichettatura SDS. L'MTSL consente di determinare la struttura e la dinamica delle proteine e di studiare le interazioni proteina-proteina e proteina-oligonucleotide.
MTSL (CAS 81213-52-7) Referenze
- Potenziali artefatti nell'uso di un sistema di proteine di fusione della glutatione S-transferasi e di metodi di risonanza elettronica paramagnetica con etichettatura di spin per studiare le interazioni proteina-proteina. | Antoniou, C. and Fung, LW. 2008. Anal Biochem. 376: 160-2. PMID: 18316034
- Conformazione dello stato di canale chiuso della colicina A nei proteoliposomi: un modello a ombrello. | Padmavathi, PV. and Steinhoff, HJ. 2008. J Mol Biol. 378: 204-14. PMID: 18353363
- Conformazione e posizione in membrana della regione che collega i due domini C2 della sinaptotagmina 1 mediante site-directed spin labeling. | Huang, H. and Cafiso, DS. 2008. Biochemistry. 47: 12380-8. PMID: 18956883
- Nuove intuizioni sul ruolo del dominio N-terminale disordinato di WIP rivelate dalla caratterizzazione strutturale NMR. | Elazari-Shalom, H., et al. 2015. FEBS J. 282: 700-14. PMID: 25495558
- Un approccio combinato NMR e computazionale per studiare il legame dei peptidi con una proteina Armadillo ripetuta progettata. | Ewald, C., et al. 2015. J Mol Biol. 427: 1916-33. PMID: 25816772
- Studio strutturale e biofisico dell'interazione di un dominio SH2 mutante di Grb2 (W121G) con il suo fosfopeptide cognato. | Papaioannou, D., et al. 2016. Protein Sci. 25: 627-37. PMID: 26645482
- Misure di distanza nella peridina-clorofilla a-proteina mediante spettroscopia PELDOR indotta dalla luce. Analisi della localizzazione allo stato tripletto. | Di Valentin, M., et al. 2016. Biochim Biophys Acta. 1857: 1909-1916. PMID: 27659505
- Regolarizzazione ottimale di Tikhonov per la spettroscopia DEER. | Edwards, TH. and Stoll, S. 2018. J Magn Reson. 288: 58-68. PMID: 29414064
- Etichettatura di spin nucleotidici per la spettroscopia ESR di proteine che legano l'ATP. | Muok, AR., et al. 2018. Appl Magn Reson. 49: 1385-1395. PMID: 30686862
- Un interruttore molecolare che regola la repressione e l'attivazione trascrizionale di PPARγ. | Shang, J., et al. 2020. Nat Commun. 11: 956. PMID: 32075969
- La preformazione strutturale a lungo raggio della proteina associata a yes precede la formazione del complesso di incontro con TEAD. | Feichtinger, M., et al. 2022. iScience. 25: 104099. PMID: 35378854
Informazioni ordini
| Nome del prodotto | Codice del prodotto | UNITÀ | Prezzo | Quantità | Preferiti | |
MTSL, 10 mg | sc-208677 | 10 mg | $170.00 | |||
MTSL, 50 mg | sc-208677A | 50 mg | $400.00 |
